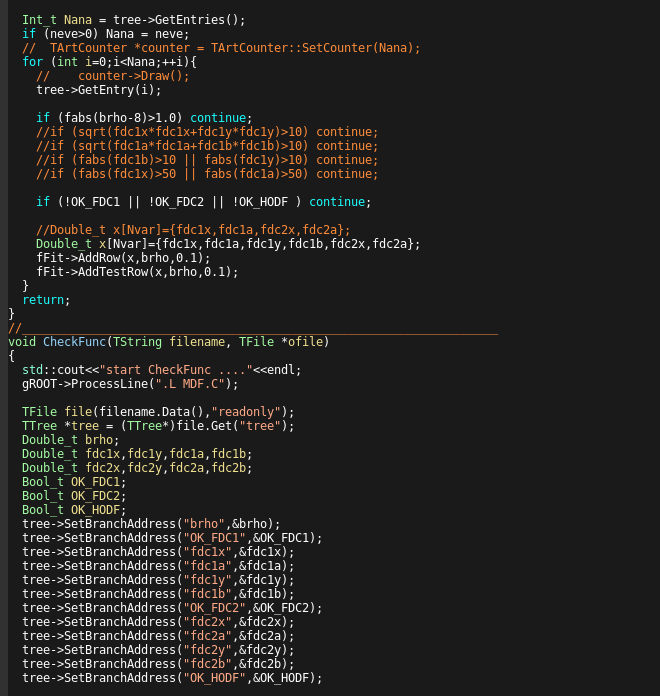
東京工業大学 >
大学院理工学研究科 >
基礎物理学専攻 >
中村研究室 >
メンバー >
栗原篤志 >
smシミュレーター 使いかた
このページでは私のわかる範囲で簡易的にsmシミュレーターの使い方について説明する。(2019/09/24)
使用するのはsmsimulator4.1だが、geant4.10では動かないらしいので注意。
geant4のインストールやsetupなどは
/smsimulator4.1/README
に書いてあるので省略する。(よく覚えていないが、
基本的にはsetup.sh内のTARTSYSを自分のanarootのsrcに書き換えて実行すれば大丈夫だと思う。)
setupまで終わったら
/smsimulator4.1/work
を自分の作業用ディレクトリとして任意の場所にコピーして作業開始。
まずはじめに
最初の最初なのでまずは動かしてみよう。
自分の作業用のwork内に入るといくつかのディレクトリがある。
私はDayone実験の解析をしているので、simdayoneに侵入する。(どれでも大差ないと思うが、
自分の解析している実験があればそれを選んだ方がいいと思う。)
また幾つかのディレクトリがあるが、一旦無視して
$ simdayone
すると、
大量の文字のあとに、
Idle>
なにか打てと言っている。(出てこない場合やsimdayoneという命令が無い場合は
setup.shやmakeの実行がうまくいってない場合が考えられる。上手くいってない場合はroot(CERN)にも入れないので、
rootと打って入れない場合はREADMEを読み返してみよう。)
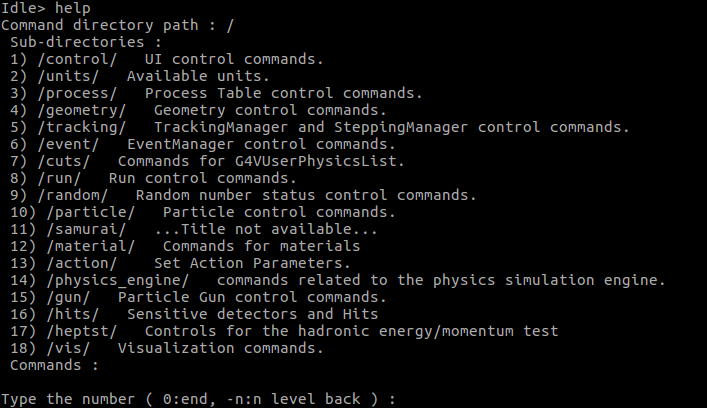
助けを求めたらヒントをくれるみたい。番号を選ぶと
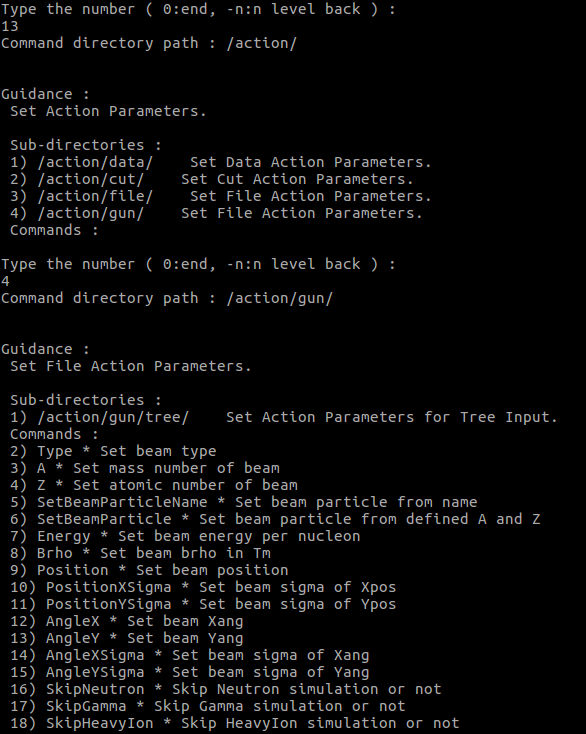
ここではbeamにする粒子の種類や位置、角度、エネルギーなどを決められるようだ。
困ったときはhelpで出来ることをチェックするのもいいかもしれない。
結局、Geant4で出来ることはこのhelpで確認出来る程度のことなので、私はこれさえ知っていればなんとかなると思っている。
さて、折角なのでこのままbeamを出してみよう。
Idle> /run/beamOn 1
と入力する。beamを1本出してという命令。
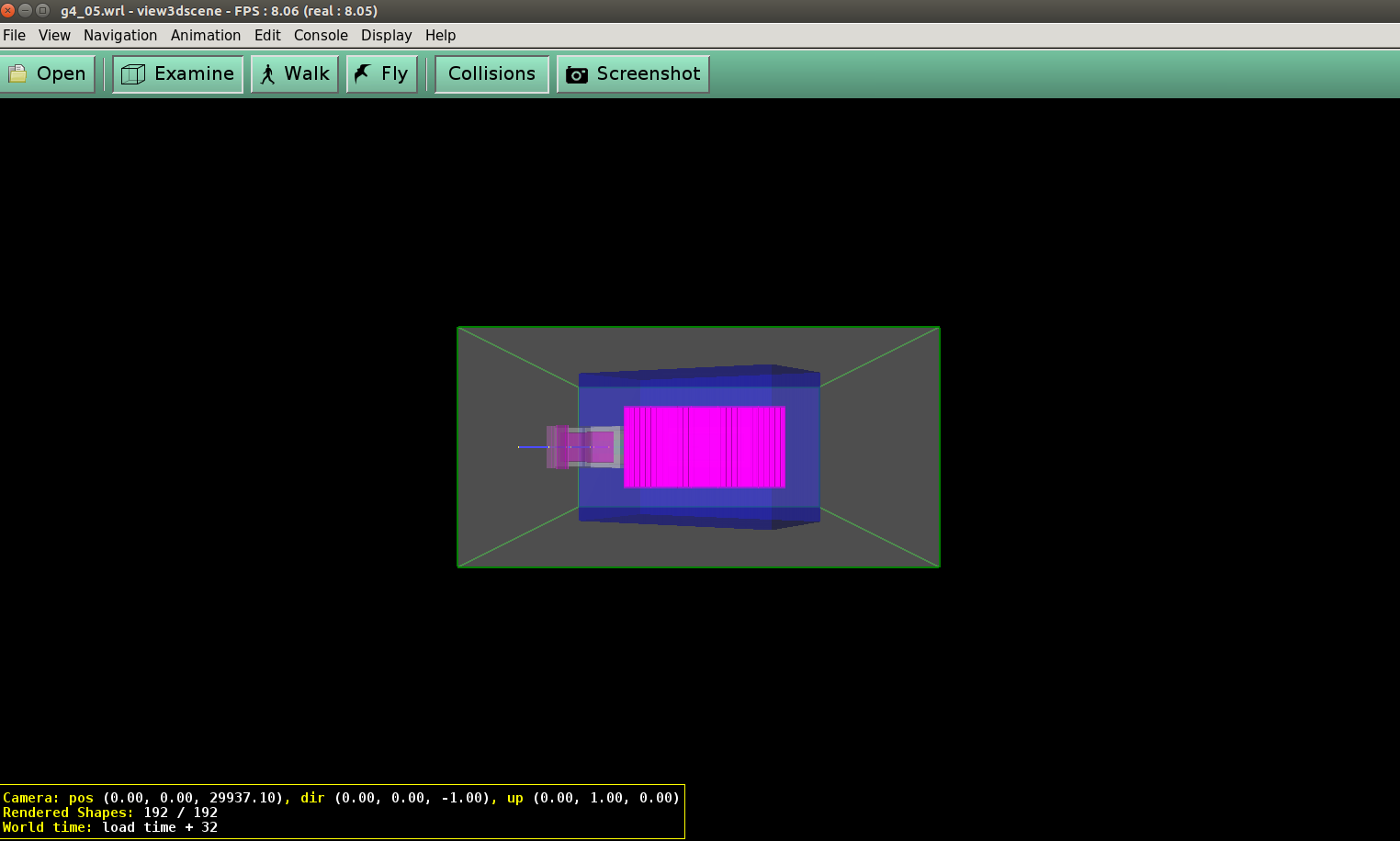
なにか出て来た。これは実はマウスのドラッグで視点を変えられる。
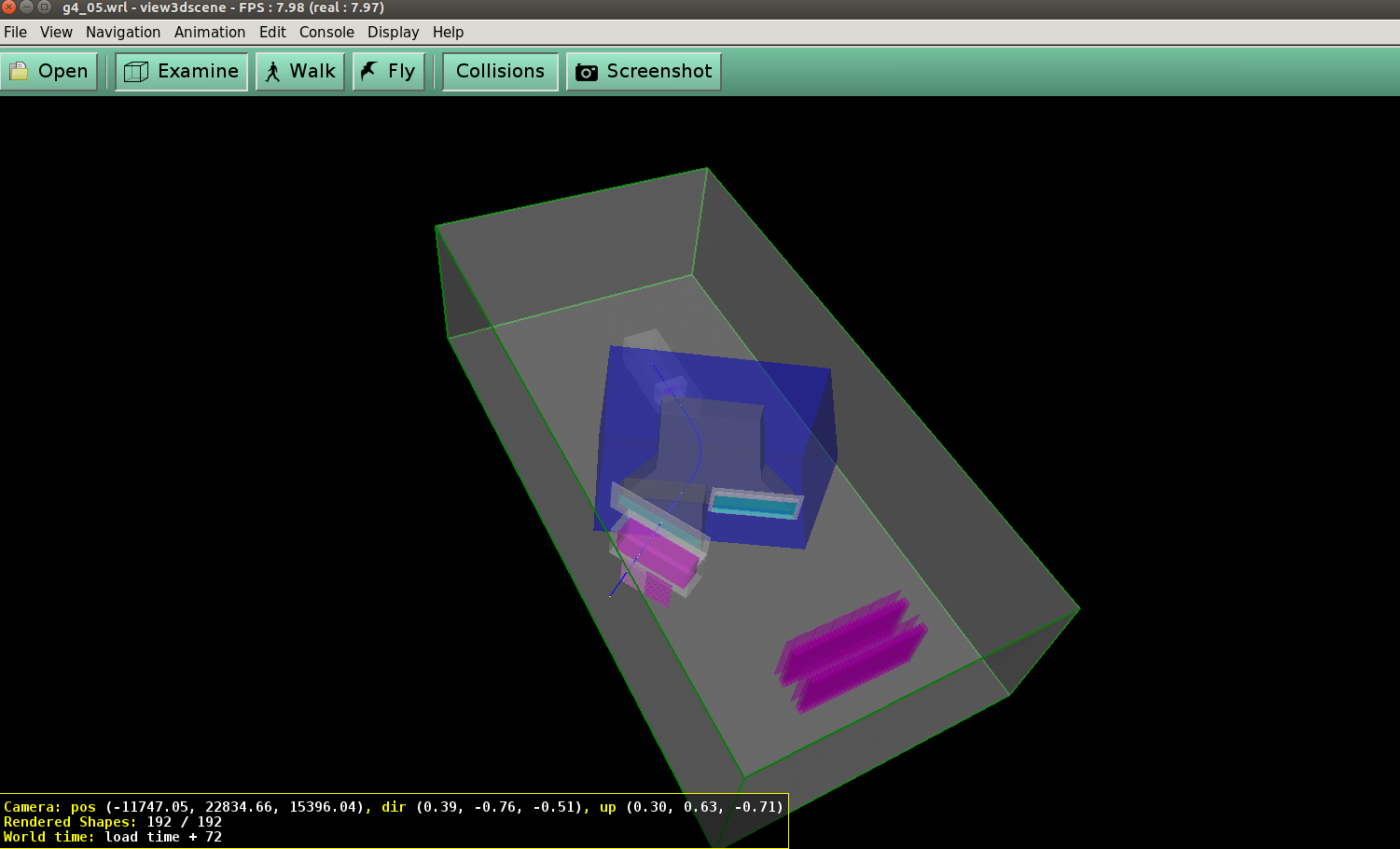
変えてみると、見たことあるようなSAMURAIが出て来た。ズームしたり、視点変えたりして遊んでるだけでもちょっとたのしい。
とまあ、とりあえず動かせることは確認できたので、今度はマクロを書いてたくさんbeamを飛ばしたり、
複雑な命令を出せるようにしよう。(|Idle>から出るときはexitと打てばよい。)
そのためにまずは作業用workディレクトリの中身を確認しよう。
ディレクトリ
geometry
その名のとおり、NEBULA HOD FDC2 SAMURAIMagnet などの配置が書いてある。
自身の実験のセットアップに合うものを探すか、改造して使ってください。
g4mac
シミュレータの制御を行う
- ビームの種類やエネルギー
- 読み込むファイルや書き出すファイル
などなど...
基本的にsimdayone上にリンクを張っておくと使いやすくなる。
$ simdayone vis.mac
などとして使う。
exampleがあるのでそれを参考に書くことをおすすめする。
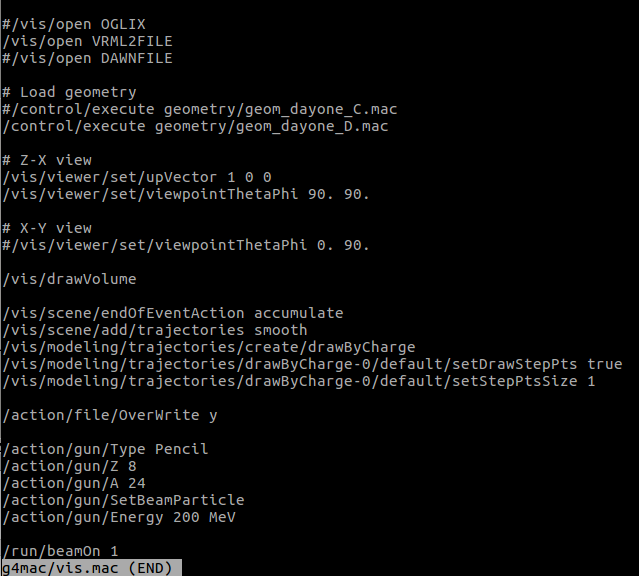
まず、左の図から説明する。
一番上の/vis/open VRML2FILE をコメントアウトするとSAMURAIエリアの図が書き出されることがなくなる。
これは複数本beamを出すときにはコメントアウトしないと非常に時間がかかるので、一粒子軌道の確認程度に使う。
/control/execute の文はセットアップに合う方を選択する。無ければ自分で細かく書く必要があり面倒。
/vis/ で始まる部分はviewerの設定なので詳しく知らない。弄ったことない。
/action/ で始まる部分はbeamの種類やエネルギーなどを決めている。
最後のbeamOnは書き加えた部分。書かないとbeamが出ないのでviewerも起動しない。
逆にviewerをONの状態でbeamOn 10000なんてやった日には一生帰って来ないので気を付けよう。
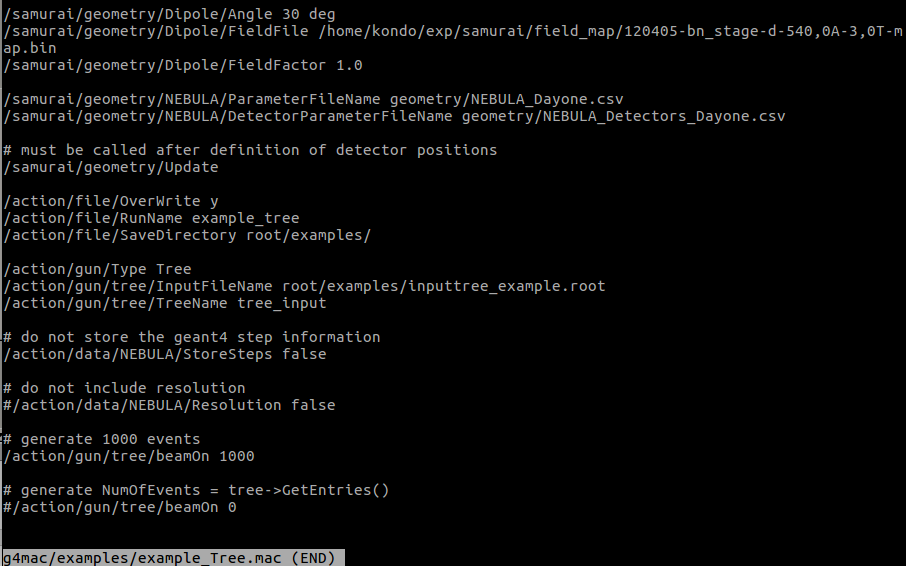
続いて、右の図の説明。
/samurai/geometry の部分はgeom_dayone_D(C).macに書いてある内容と同じなので、代用可能。
逆に、丁度いいセットアップがない人はこうやって書く。
/action/file/ の部分は生成するファイル名を書いている。
example_tree0000.rootというファイルがsimdayone上のroot/example/に作られる。
inputfileで粒子の条件などが決められているので、beamOnとしても条件にあわせてシミュレーションが行われる。
macros
名前の通り、inputfileを生成したり、outputfileを解析したりするマクロがおいてある。
ここからはマクロの基本的な書き方について説明する。
inputの生成とoutputの解析
大量のビームを生成する場合、
rootを用いてinputfile(tree)を生成
↓
vis.macで制御して、simulationを行う(outputfile(tree)を取得)
↓
rootを用いてoutputfileを解析
というのが、簡単な流れである。
まずは、inputfileの生成から説明する。
やはりここにもexampleが用意されているので、それを用いて説明していく。
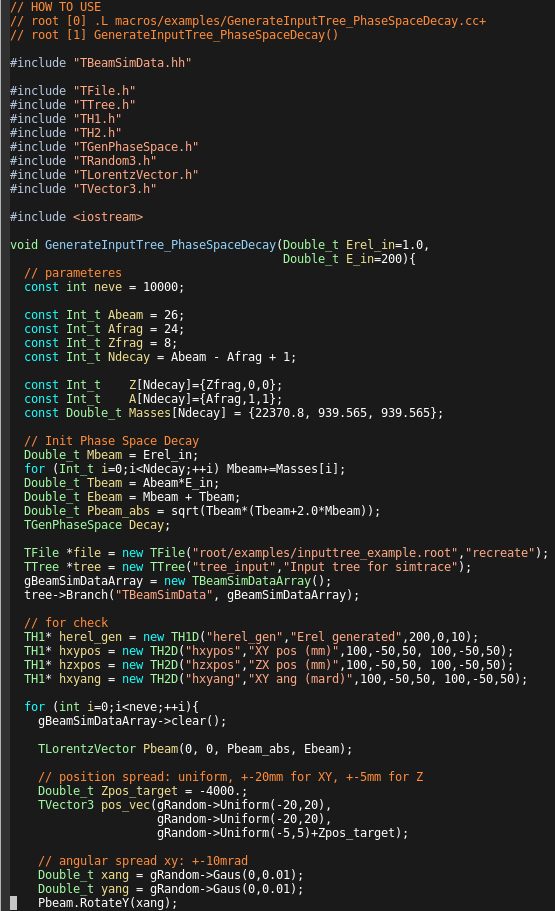
- " #include TbeamSimData.hh "
smsimulatorを使ううえで必ず必要になってくるのがTBeamSimDataというクラス。
TBeamSimDataというクラスは粒子の陽子数Z,核子数A,四元運動量ベクトル,位置ベクトルからなる。
このTBeamSimDataを生成する各粒子について定義し、
TBeamSimDataの集合(vector)であるTBeamSimDataArray(ここではgBeamSimDataArrayという名前)に格納する。
このTBeamSimDataはヘッダーファイルであるTBeamSimData.hhにおいて、
extern TBeamSimDataArray* gBeamSimDataArray;
と書かれており、includeするだけでgBeamSimDataArrayが使えるようになっている。
粒子のZ,Aの決定。
粒子のエネルギーを決定。
生成ファイル名。inputfileの名前になるので、当然g4macのinputfileと同じにしておく。
- " gBeamSimDataArray = new TBeamSimDataArray(); "
初期化。これをしないと、treeに詰めたときに値がバグる。
また、fileの中身はgBeamSimDataArrayのみをBranchにもつtreeで良い。
gBeamSimDataArrayはTBeamSimDataのポインタで定義されているので(??)
treeに詰めるときgBeamSimDataArrayの前に& は付けない。
(TClonesArrayやTVector3、TLorentzVectorはポインタのポインタを詰める必要があるが、
vectorはそのアドレスを詰めるんだったと思う。書いててわかんなくなってきた。)
ほかのBranchを追加したらどうなるかは確認してない。
beamのスタート位置は直方体の一様分布。
発射角度は球状の正規分布。
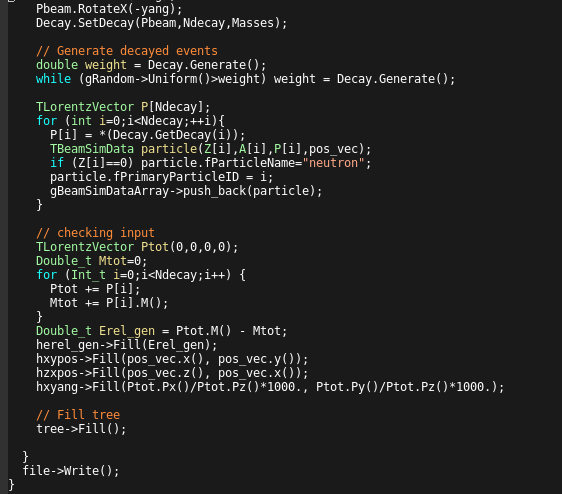
TGenPhaseSpace::SetDeCay()はBool_tの関数で、不適切な値を代入するとfalseを返してくれる。
falseを返す場合、四元運動量から求めた質量と代入した質量が一致しない、などの場合がある。(筆者はそれで一度手間取った。)
また、SetDecayに代入する質量や運動量はtargetでの反応後の粒子のものなので、間違えないように。
逆に、GetDecayから得られる値はPhaseSpaceDecayをした後の運動量で、これをsimulationで飛ばしてあげる。
TBeamSimDataに必要な値を詰めて、順にgBeamSimDataArrayに詰めていく。
あとは詰めた値を確認用のヒストグラムに詰める。
もし、実験の分布を使いたいのであれば、TH1::GetRandom()やTH2::GetRandom2(double &x,double &y)を用いると良い。
これはhistogramの形に応じてランダムな値を返してくれる関数で、何度も値を取ってきて、
それをhistogramに詰めなおすと、元の分布と同じかたちの分布ができる。
TH1::GetRandom()は返り値を代入すればいいが、TH2::GetRandom2(double &x,double &y)
は()内の変数に値を代入するもので、使いかたが違うので注意してほしい。
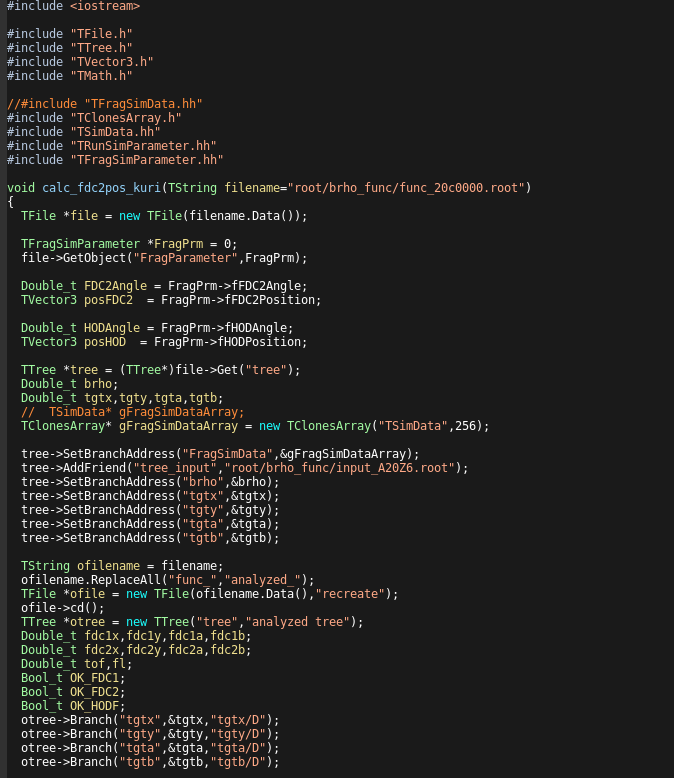
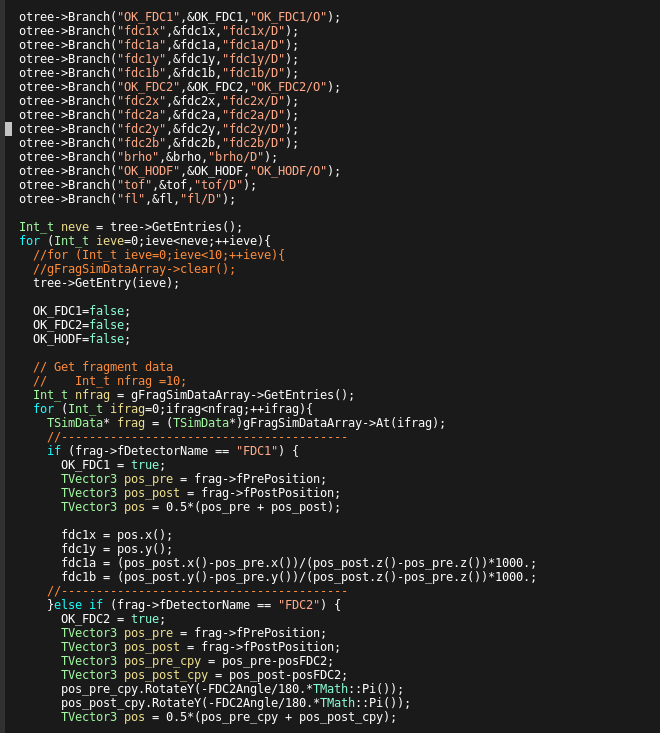
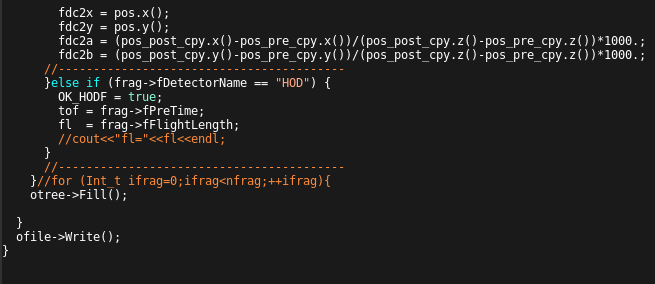
続いては、outputfileの説明に移る。
outputfileのtreeには大きく分けて4種類のBranchがある。
- beamの情報(TBeamSimData)
- fragmentの情報(TFragSimData)
- neutronの情報(TArtNEBULAPla)
- FDCの情報
出来たrootfileの中身を確認すると100個近いBranchが現れるが大まかにはこの4種類しかない。
取り出す書き方などを一度見てしまえば、あとはいつものtreeを解析するだけでいいのでexampleを見ていこう。
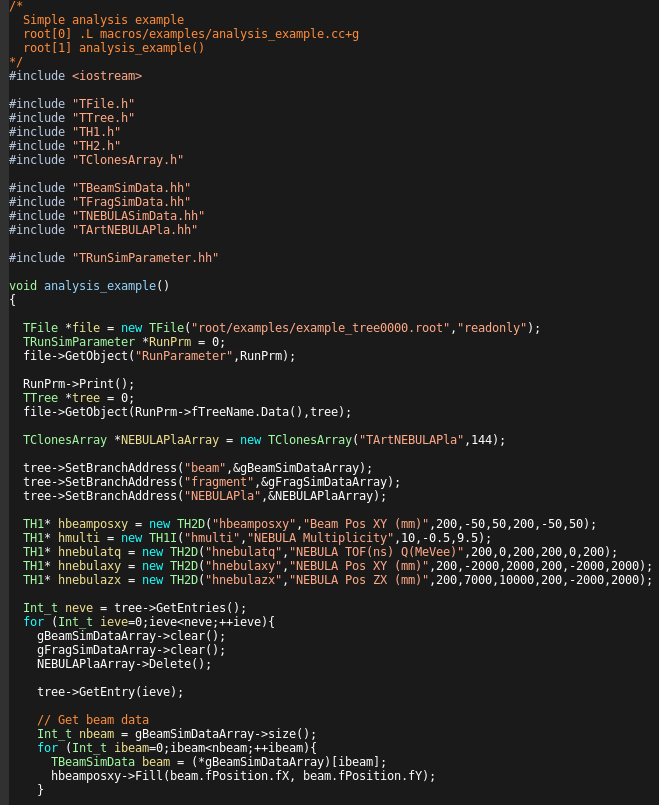
正直、取り出し方はこうです。以上の解説など無いが、このままこのマクロを実行しても(私の環境では)正常に動作しなかったので、
修正案だけメモしておく。
私の環境で動作しなかったのはTFragSimData.hhとTNEBULASimData.hhが存在しないから
(このexampleでもTNEBULASimDataは必要だったのかは謎)なので、includeの部分をコメントアウトする。(TBeamSimData.hhは残す)
そして代りに使用するTSimData.hhとTFragSimParameter.hhをincludeする。
TRunSimParameterと同じような書き方でTFragSimParameterは使用する。
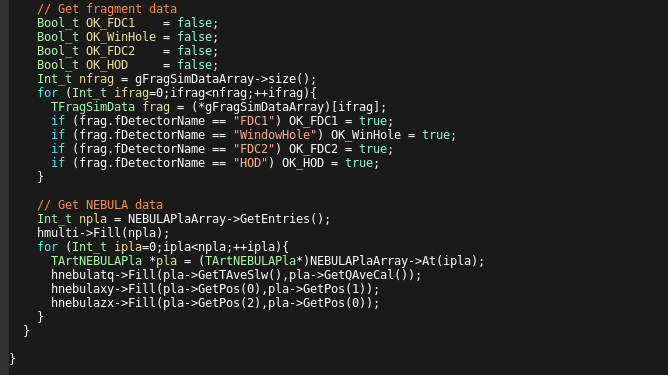
TFragSimData.hhをincludeしていないのでgFragSimDataArrayは使えないから、
自分でTSimDataのクラスのTClonesArrayを定義する。
(名前はgFragSimDataArrayにすると後の変更が少くて済む。書き方はNEBULAPlaArrayを真似するといい)
これだけやってしまえば基本的にはTSimDataとTFragSimDataの使いかたは同じなので、自身で細かい部分は修正して欲しい。
一応、私が使っているコードの余計な部分を除いたものを
ここ
に置いておく。
説明などは勘弁。また、そのまま写しただけだと(省いた部分のせいで)動かない可能性があるので、参考程度に。
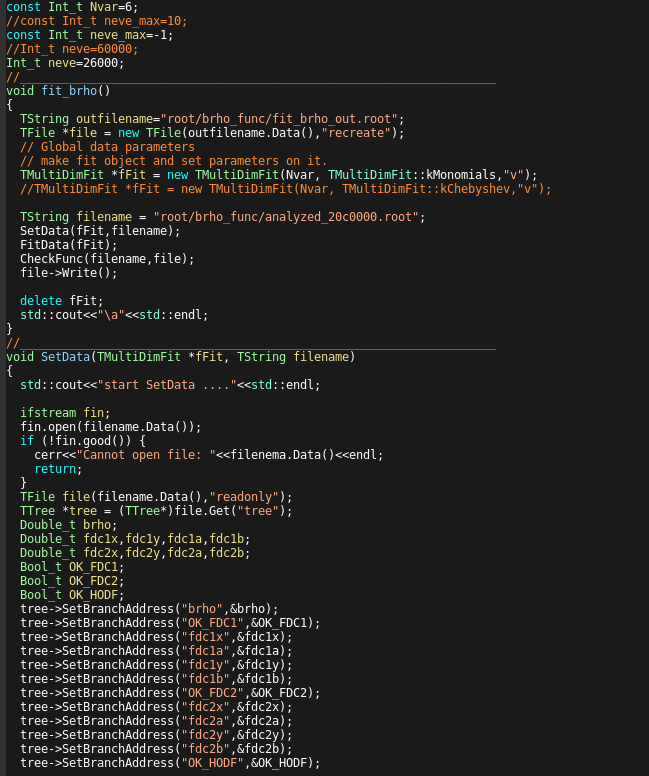
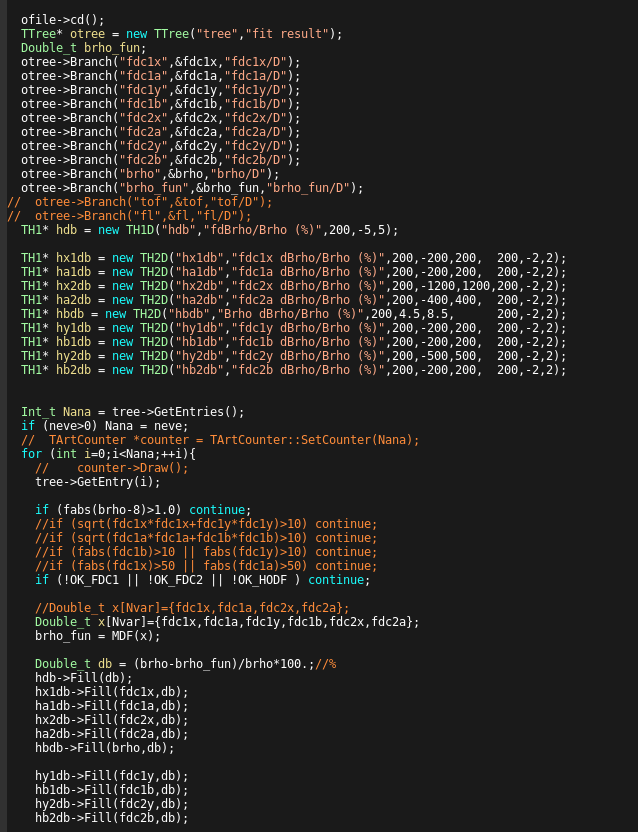
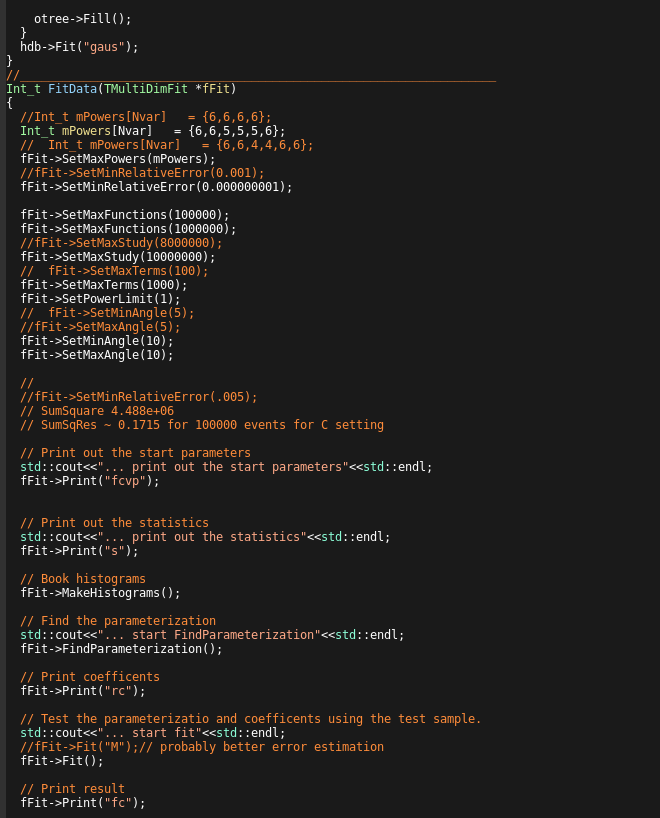
下流Bρの決定
ここからは、smsimulatorを使う目的の一つである、下流Bρの決定について説明する。
そもそも下流Bρをなぜシミュレーションで求めるのかというと、
下流のfragmentはSAMURAI Magnetで曲げられており、Flight Lengthが決められないからである。
そこでsimulationを使って、あるBρの粒子がFDC1のどこにどの角度で入射したらFDC2のどこにどんな角度で到達する、
ということを1つ1つ調べて多変数関数にしてしまおう、
というのが下流Bρの決定にsimulatorを使うことになった経緯である。
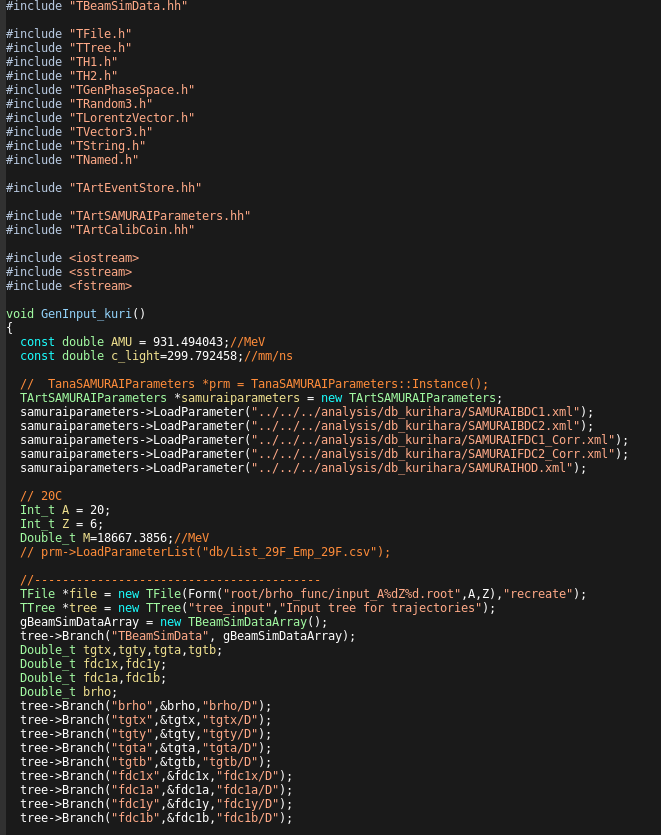
こちらも基本的には説明は不要であると考えている。
関数名が変なのは、アイデンティティなので多めに見てほしい。
いつものように、parameterを読み込んでファイルを生成してBranchをつくる。
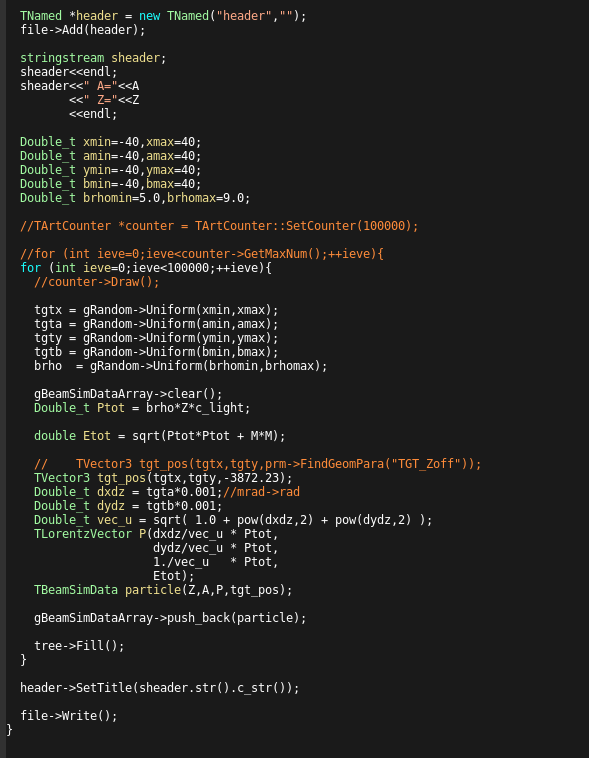
ここではターゲットの分布の上限と下限を決めている。
決めた上限下限に従って、一様分布で値をランダムに決める。
この分布が実験の分布を含んでいると分解能が上がる。
というか、含めるのは難しくないので、ある程度大きく作るのがおすすめ。
あとは、位置と角度、エネルギーから運動量を決定し、前のexampleと同じようにTBeamSimDataArrayに詰めていく。